Nature Communications:西湖大学马丽佳团队开发新型CRISPR脱靶和DNA易位检测工具
时间:2022-12-15 11:59:50 热度:37.1℃ 作者:网络
基于CRISPR的基因组编辑在生物学研究和临床应用方面都显示出了巨大潜力。与小分子药物和抗体药物相比,CRISPR能够直接靶向特定核酸序列,很大程度上解决了不可成药靶点限制,具有独特优势。
CRISPR-Cas基因编辑依赖于向导RNA(gRNA)的识别和靶向,gRNA的非特异性靶向可能会引入意料之外的脱靶编辑,从而导致细胞遗传毒性。这也是CRISPR基因编辑疗法临床应用中面临的一大限制因素。因此,人们迫切需要了解脱靶编辑的结果,以及由此产生的DNA易位(DNA translocation)等问题。
DNA易位一直是CRISPR基因编辑的一个重要问题,尽管它发生的频率相对较低,但通常会导致高遗传毒性。DNA易位的潜在风险通常发生在例如使用CRISPR基因编辑的CAR-T细胞中,多个gRNA被引入到T细胞中进行多位点基因编辑,导致DNA双链断裂(DSB)末端的易位风险。随着基于CRISPR基因编辑的同种异体CAR-T细胞疗法开始进入临床试验,迫切需要开发出能够系统性鉴定DNA易位的方法。
2022年12月12日,西湖大学生命科学学院马丽佳团队在 Nature Communications 期刊发表了题为:PEAC-seq adopts Prime Editor to detect CRISPR off-target and DNA translocation 的研究论文。
该研究利用先导编辑(Prime Editor,PE)的序列插入能力,用Cas9代替PE中的Cas9n,与逆转录酶MMLV融合,开发了一种新型脱靶鉴定方法——PEAC-seq(Prime Editor Assisted off-target Characterization)。
PEAC-seq提供了一个全面和简化的策略来鉴定细胞和体内的CRISPR脱靶编辑,以及Cas9导致的DNA易位。这项技术进一步丰富了用于评估CRISPR在基础研究和临床应用中的遗传毒性的工具包。

由于CRISPR基因编辑技术对遗传疾病、癌症等疾病治疗有着很大潜力,因此对体内CRISPR基因编辑的脱靶鉴定和相应的毒性评估具有很高的要求。
2019年,刘如谦(David Liu)团队在 Nature 发表论文,开发了一种新型基因编辑工具——Prime Editor,Prime Editor在Cas9酶和gRNA两方面进行了重大改造,在gRNA的3'末端增加了一段RNA序列,形成所谓的pegRNA;将Cas9切口酶(Cas9n,只切断含PAM序列DNA单链)与逆转录酶融合,形成融合蛋白。
pegRNA的3'端序列有双重角色,一段序列作为引物结合位点(PBS),与断裂的靶DNA链3'末端互补以起始逆转录过程,另一端序列则是逆转录模板(RT模板),其上携带有目标点突变或插入缺失突变以实现精准的基因编辑。

Prime Editor的基因编辑原理
为了兼容对体内CRISPR基因编辑的脱靶检测,检测方法应该简化编辑和脱靶富集过程,而不依赖于外源片段。为此,研究团队使用Cas9代替Cas9n,与逆转录酶MMLV的融合蛋白(Cas9-MMLV),以利用Prime Editor的序列插入能力。
Cas9-pegRNA在基因组的上靶和脱靶位点都能够产生DNA双链断裂(DSB),标记序列将通过pegRNA的逆转录引入DSB位点,并通过DNA修复来插入基因组中。研究团队设计了一个21nt的插入标签,利用pegRNA模板插入标签序列进行富集。
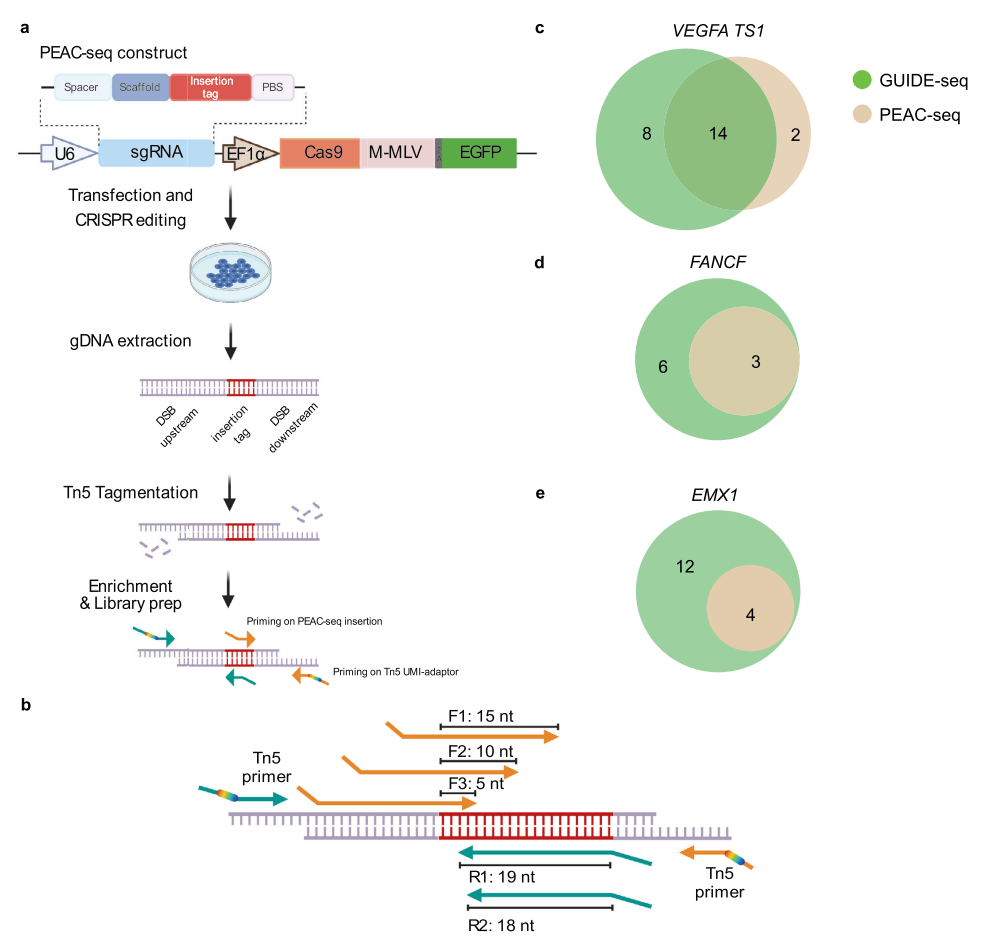
PRAC-seq技术
在PEAC-seq中,优化后的逆转录模板(RT模板)可以整合到靶位点或者脱靶位点中,进一步对这些位点进行富集。PEAC-seq伴随着CRISPR编辑和插入标签的过程,确保了编辑与PEAC-seq信号的一致性。
将PEAC-seq在细胞和小鼠体内的若干位点分别进行测试,并与GUIDE-seq、DISCOVER-seq、WGS和Amplicon-seq等检测结果进行比较,证明了PEAC-seq可以有效地识别脱靶位点。
此外,得益于定向插入的PEAC-seq标签,PEAC-seq能够系统识别DNA易位,之前的方法只能知道一些固定位点的染色体易位,而无法进行系统评估,而DNA易位通常意味着更大的细胞毒性。
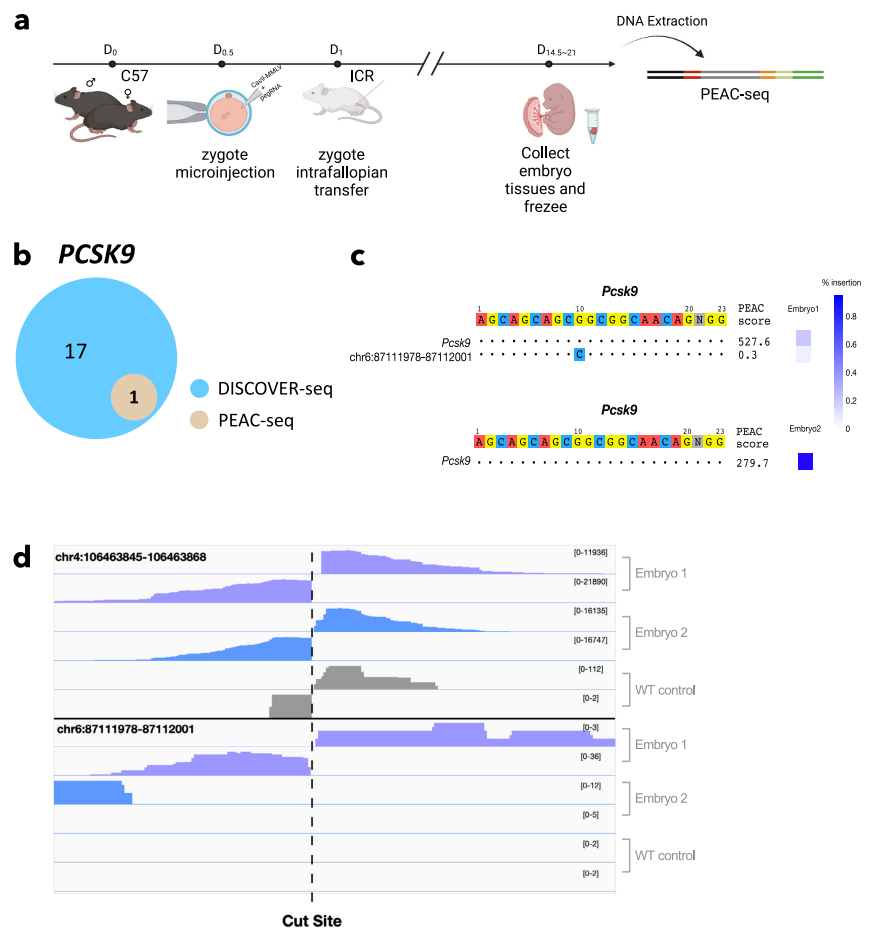
PEAC-seq从编辑过的小鼠胚胎中鉴定出PCSK9脱靶
总的来说,PEAC-seq是一种识别CRISPR脱靶和脱靶相关DNA易位的无偏倚方法。由于无需添加高摩尔浓度的外源性短双链寡核苷酸(dsODN),因此在体内CRISPR编辑中,PEAC-seq在识别脱靶和DNA易位方面具有巨大潜力,这对CRISPR基因编辑的转化应用尤其有价值。
原始出处:
Yu, Z., Lu, Z., Li, J. et al. PEAC-seq adopts Prime Editor to detect CRISPR off-target and DNA translocation. Nat Commun 13, 7545 (2022). https://doi.org/10.1038/s41467-022-35086-8.


