NEUROPATHOLOGY AND APPLIED NEUROBIOLOGY:肌萎缩侧索硬化症中外周血的RNA测序揭示了不同的分子亚型
时间:2023-12-09 17:45:24 热度:37.1℃ 作者:网络
肌萎缩侧索硬化症(ALS)是一种由大脑和脊髓运动神经元逐渐丧失引起的成人发病的神经退行性疾病。虽然外周血不是ALS疾病的主要部位,但其可及性使其成为寻找疾病生物标志物的主要目标。已经报告了几种基于血液的肌萎缩侧索硬化症的生物标志物,包括神经丝、免疫细胞(如改变的T调节细胞)、细胞因子(如IFN-γ、白细胞介素)、微RNA(如miR206)和代谢物(如血清肌酐、谷氨酸、尿酸)。

其中几种生物标志物也在脑脊液(CSF)中报告。虽然与中枢神经系统(CNS)更近,但获得脑脊液具有高度侵入性,在整个疾病进展过程中很难连续采样。单独使用时,ALS的单个候选生物标志物表现出可变的临床效用,并且缺乏一致的交叉验证。缺乏用于患者诊断、预后、队列分层和监测治疗反应的强大生物标志物是开发有效肌萎缩侧索硬化症有效疗法的关键限制因素。
改善ALS生物标志物性能的拟议方法是结合多个基于血液的生物标志物,或将它们与遗传风险因素、神经成像或电生理学等其他模式的生物标志物配对。另一种越来越可行的方法是通过将肌萎缩侧索硬化症患者生物标本应用生物特征来检查更全球性的分子特征。转录组学特别适合捕捉疾病条件下生物途径的全球快照,一些研究已利用基因表达微阵列或RNA测序(RNA-seq)来描述ALS患者全血或外周血单核细胞中的基因表达。
肌萎缩侧索硬化症人群研究的一个重要考虑因素是高度的临床和病理异质性,这可能会使检测与对照个体的差异复杂化。在检查高维数据(如RNA-seq)时,这一点至关重要,在高变量数据中需要大样本量来检测患者子群体。迄今为止报告的最大肌萎缩侧索硬化症对照血液基因表达数据集包括397名肌萎缩侧索硬化症患者、645名对照组和75名由微阵列评估的肌萎缩侧索硬化症模拟者。该数据集随后的重新审讯报告了一个高准确诊断模型、患者亚群和预测患者生存的基因小组;然而,这些发现仍有待独立队列验证。
2023年10月11日发表在NEUROPATHOLOGY AND APPLIED NEUROBIOLOGY的文章,描述了一个由96例零星ALS(sALS)病例和48个匹配的对照参与者组成的外周血RNA-seq数据集。我们进行了全面分析,以确定ALS患者诊断、预后和分层的生物标志物候选者。这首次确定了ALS患者和对照组之间的基因和转录水平表达差异。
研究人员使用机器学习来开发准确的诊断模型(ALS与对照组),但报告称,全血基因表达对于预测包括生存在内的患者表型的信息较少。最后,本文采用无监督聚类分析来区分具有独特基因表达特征和预测免疫细胞比例的肌萎缩侧索硬化症患者亚群。该RNA-seq数据集是公开可用的,可以在GSE234297下的NCBI基因表达综合(GEO)访问。
为96例澳大利亚零星ALS(sALS)病例和48名健康对照组(NCBI GEO加入GSE234297)生成了全血RNA-seq数据。评估了sALS控制基因表达、转录物使用和预测白细胞比例的差异,并使用路径分析来预测生物过程的活动状态。加权基因共表达网络分析(WGCNA)和机器学习算法应用于搜索诊断和预后基因表达模式。采用无监督聚类分析来确定是否可以检测到SALS患者亚组。
研究结果显示,与对照组相比,在SALS患者中发现了245个差异表达的基因,丰富了免疫、代谢和压力相关途径。SALS患者还展示了一小部分基因在转录使用方面的开关。本文建立了一个分类模型,使用20个基因的表达将ALS患者与准确率为78%(敏感性:79%,特异性:75%)的对照组区分开来。聚类分析确定了四个患者亚群,其基因表达特征和免疫细胞比例反映了不同的外周效应。
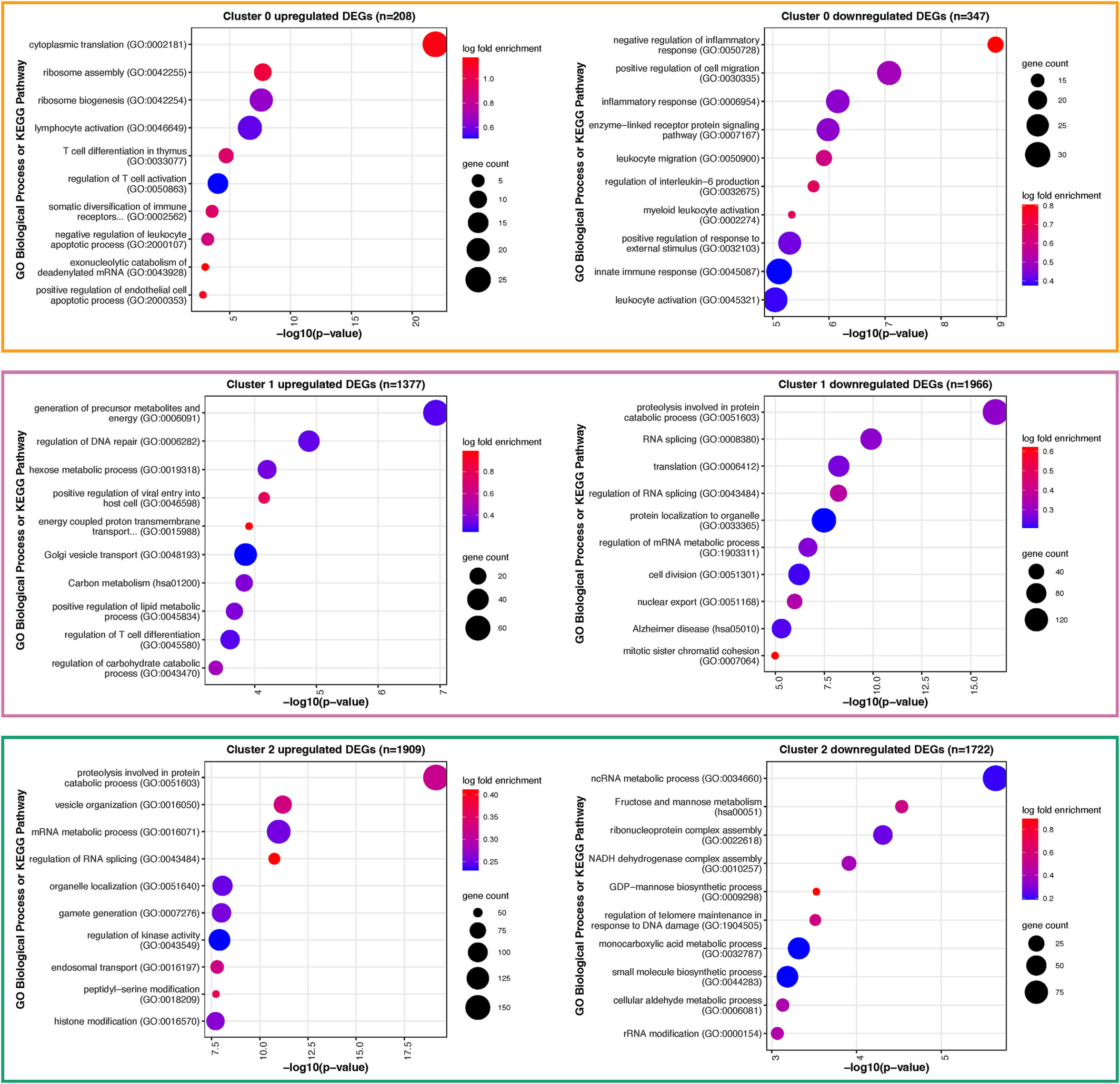
GO生物过程和KEGG途径丰富了在sALS亚群中显著上调或下调的基因
综上所述,本文对独立的ALS对照外周血RNAseq队列进行了全面分析,并强调了不同患者亚群的存在。观察到的ALS血液基因表达谱中的异质性使诊断和预后生物标志物发现复杂化,未来的RNA-seq研究应采用纵向样本和/或中枢神经系统血液匹配队列。
原文出处
, , , et al. RNA sequencing of peripheral blood in amyotrophic lateral sclerosis reveals distinct molecular subtypes: Considerations for biomarker discovery. Neuropathol Appl Neurobiol. 2023; 49(6):e12943. doi:10.1111/nan.12943


