Nature Methods:南京大学黄硕团队开发了新的蛋白质测序方法
时间:2023-09-27 14:16:56 热度:37.1℃ 作者:网络
天然蛋白质由20种蛋白质原性氨基酸及其翻译后修饰(PTMs)组成。然而,由于缺乏一种合适的纳米孔传感器,可以同时区分所有20种氨基酸及其PTMs,因此具有纳米孔的蛋白质直接测序尚未实现。
2023年9月25日,南京大学黄硕团队在Nature Methods 在线发表题为“Unambiguous discrimination of all 20 proteinogenic amino acids and their modifications by nanopore”的研究论文,该研究提出了一个工程化的异八聚耻垢分枝杆菌孔蛋白A (MspA)纳米孔(含有一个单一的Ni2+修饰)。它可以完全识别所有20种蛋白质原性氨基酸和4种具有代表性的修饰氨基酸,即Nω,Nω-二甲基精氨酸(Me-R), O-乙酰-苏氨酸(Ac-T), N4-(β-N-乙酰-D-氨基葡萄糖)-天冬酰胺(GlcNAc-N)和O-磷酸丝氨酸(P-S)。
在机器学习的辅助下,准确率达到了98.6%。氨基酸补充片和肽酶消化的氨基酸也用这种方法进行了分析。这种同时识别所有20种蛋白质原性氨基酸及其PTMs的能力表明,使用这种基于纳米孔的策略可以实现蛋白质测序。

蛋白质是生命活动的重要执行者,但只有少数技术,如Edman降解和质谱,有能力确定蛋白质的氨基酸序列。蛋白质测序的检测限制也阻碍了低丰度蛋白质的表征。单分子蛋白测序仪可以提高翻译后修饰(PTMs)的灵敏度和信息。纳米孔是一种多用途的单分子传感器,在核酸测序方面取得了显著进展,已成为一种有前途的候选传感器。
尽管在实现蛋白质的纳米孔易位方面做出了巨大的努力,但无法仅从不受控制的蛋白质易位中获得序列信息。根据纳米孔诱导相移测序(NIPSS)策略,可以通过纳米孔扫描肽-寡核苷酸偶联物,以报告包含序列依赖肽信息的痕量特征。然而,这种方法仍然受到纳米孔分辨率的阻碍,由于20种蛋白质原氨基酸序列组合的复杂性,纳米孔分辨率不足以可靠地解码蛋白质序列。
另一种方法是通过水解方法对蛋白质进行测序,其中肽酶消化的氨基酸通过纳米孔顺序读取,类似于用蛋白酶体纳米孔所演示的。然而,这需要一个纳米孔,它可以明确地识别所有的蛋白质生成氨基酸及其PTMs,这还没有实现。在此之前,一个CuII-phenanthroline修饰的α-溶血素(α-HL)纳米孔已经被证明可以直接识别5对氨基酸对映体。然而,由于α-HL的分辨率不足,该方法无法同时区分所有20种氨基酸。气溶酶纳米孔也被用于识别含有单个末端氨基酸差异的八肽。这种方法的分析物是短肽,而不是独立的氨基酸,据说20种这种肽中只有13种被鉴定出来。其他的纳米孔氨基酸鉴定方法也有报道,但直接鉴定全部20种蛋白质原性氨基酸仍未实现。单分子氨基酸的识别可以通过识别隧道进行,但目前报道的事件识别仍不能令人满意。
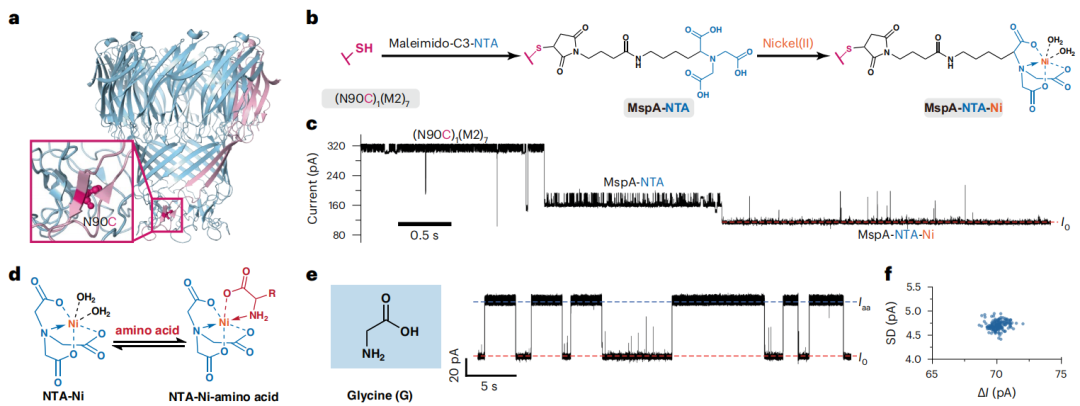
用于氨基酸传感的Ni-NTA修饰纳米孔的构建(图源自Nature Methods)
耻垢分枝杆菌孔蛋白A (MspA)是一种锥形的生物纳米孔,广泛应用于核酸的纳米孔测序。经过改造的MspA还可以用作纳米反应器,用于监测单分子化学反应。离子和小分子,如四氯金酸盐(III)、神经元递质、抗COVID-19药物、儿茶酚胺对映体、单糖、单磷酸核苷和糖醇,已经使用含有合适反应适配器的MspA纳米孔进行了鉴定。在固定化金属亲和色谱(IMAC)中,镍-硝基三乙酸(Ni-NTA)亲和柱用于纯化含有六组氨酸标签的重组蛋白,受此启发,该研究设计并制备了一种在孔缩处含有单一Ni-NTA适配器的异八聚MspA,用于氨基酸传感。虽然Ni-NTA修饰应用于固态纳米孔的整个内腔中,用于检测组胺和His标记的蛋白质,但迄今为止尚未报道含有单一Ni2+修饰的生物纳米孔。
该研究提出了一个工程化的异八聚耻垢分枝杆菌孔蛋白A (MspA)纳米孔(含有一个单一的Ni2+修饰)。它可以完全识别所有20种蛋白质原性氨基酸和4种具有代表性的修饰氨基酸,即Nω,Nω-二甲基精氨酸(Me-R), O-乙酰-苏氨酸(Ac-T), N4-(β-N-乙酰-D-氨基葡萄糖)-天冬酰胺(GlcNAc-N)和O-磷酸丝氨酸(P-S)。
在机器学习的辅助下,准确率达到了98.6%。氨基酸补充片和肽酶消化的氨基酸也用这种方法进行了分析。这种同时识别所有20种蛋白质原性氨基酸及其PTMs的能力表明,使用这种基于纳米孔的策略可以实现蛋白质测序。
参考消息:
https://www.nature.com/articles/s41592-023-02021-8


