IVD前沿丨食管鳞状细胞癌及癌前病变早筛新技术
时间:2024-05-28 22:00:33 热度:37.1℃ 作者:网络
食管癌(EC)是最常见的胃肠道恶性肿瘤之一,食管鳞状细胞癌(ESCC)是EC的主要组织学亚型,约占新发EC病例的88%,主要发生在东亚和中亚。ESCC预后不佳,其特点是5年总生存率低(美国和中国分别为18.5%和36.9%),这主要归因于晚期诊断。相比之下,早期ESCC通过内镜整体切除可达到近100%的5年疾病特异性生存率,无需系统治疗。因此,早期发现对于提高ESCC患者的生存和生活质量至关重要。
诊断ESCC及其前驱病变的金标准仍然是碘染色内镜检查。然而,内窥镜筛查的广泛采用面临着挑战,包括低依从性和大规模人群进行内窥镜检查的巨额费用。液体活检方法能够检测血浆中游离细胞DNA (cfDNA)中的循环肿瘤DNA (ctDNA),为非侵入性早期癌症检测提供了一条有前途的途径。然而,很少有研究评估液体活检在ESCC诊断中的应用。
近日,杂志Nature Communications上发表了一篇题为“Multimodal analysis of cfDNA methylomes for early detecting esophageal squamous cell carcinoma and precancerous lesions”的文章,作者开发了扩展多模态分析(EMMA)方法,同时识别cfDNA样本全基因组亚硫酸盐测序(WGBS)数据中与癌症相关的差异甲基化区域(DMRs)、CNVs和片段化特征。EMMA显著提高了检出率,AUC为0.99,在验证队列中检测到87%的ESCC和62%的癌前病变,特异性>95%。证明了cfDNA甲基化组学的多模态分析在ESCC早期检测和分子特征监测中的潜力。

图片来源:Nature Communications
主要内容
cfDNA全基因组亚硫酸盐测序中的EMMA框架概述
为了提高ESCC的早期检测,作者开发了基于“组织- cfDNA-组织”策略的EMMA框架(下图a),全面分析cfDNA WGBS数据中的癌症衍生的差异甲基化区域(DMRs)、拷贝数变异(CNVs)和片段特征。肿瘤来源的DMRs和CNVs是从155例ESCC患者的原发肿瘤和配对组织中的WGBS和WGS数据确定的。随后,在cfWGBS数据中检测了ESCC衍生的DMRs和CNVs,以及短cfDNA片段大小的比例(FSRs)来训练诊断模型。在外部ESCC队列和癌前病变队列中独立评估了每个诊断模型的性能。
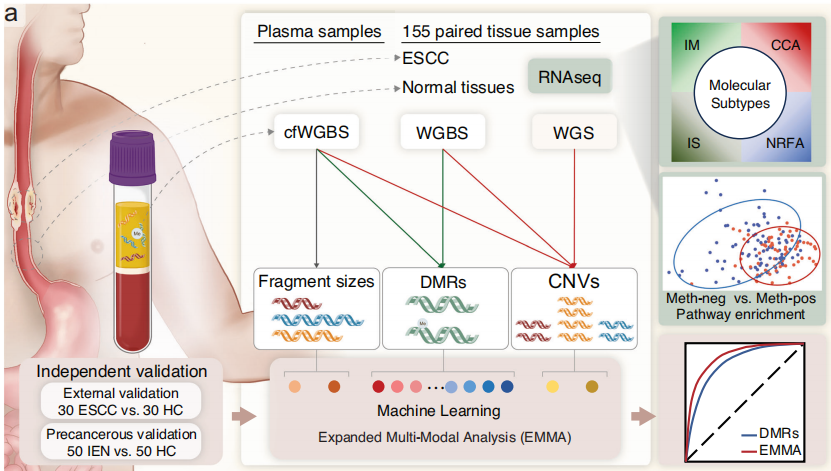
cfDNA全基因组亚硫酸盐测序中的EMMA框架概述。
图片来源:Nature Communications
cfDNA甲基化标记的识别与性能分析
在cfDNA中,作者通过比较150名ESCC患者和150名HC的WGBS数据,确定了650个DMRs。随后计算每个样本中每个DMR的cfDNA恶性比率,并利用随机森林算法生成了预测模型(ESCC-cfMeth评分),使用50个DMR的cfDNA恶性比率构建(图a),包括40个低DMR和10个高DMR。在外部验证队列中,AUC为0.89,在癌前验证队列中,AUC达到0.87 (图 b)。
作者注释了这50个DMRs的基因,有部分基因在ESCC发育中起关键作用。综上所述,这些发现不仅强调了这些cfDNA甲基化特征在ESCC诊断中的可靠性和可解释性,而且表明了它们在ESCC进展中的潜在功能相关性。
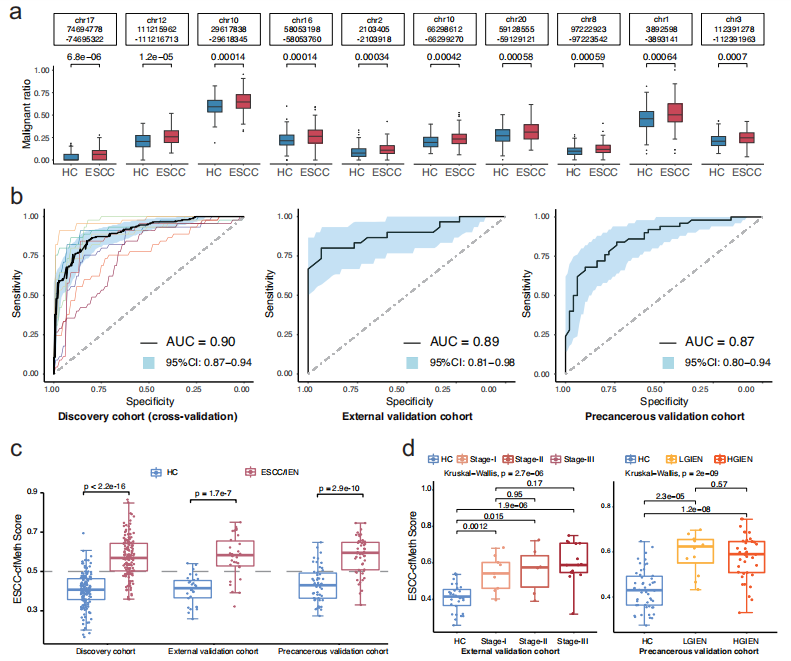
cfDNA甲基化标记的识别与性能分析。
图片来源:Nature Communications
用于ESCC检测的cfDNA CNVs识别
作者开发了一种基于WGBS的方法来识别组织和cfDNA样本中的共同的CNVs。与HC的CNV事件相比,ESCC患者中有153个区域的CNV事件发生率明显更高,其中111个扩增区域和42个缺失区域。从HGIEN开始,CNV事件的发生率逐渐增加,并与癌症分期和分级呈正相关(图d)。这些结果突出了cfDNA中CNV事件的高特异性(范围从98%到100%),并且它们与肿瘤进展和晚期疾病呈正相关。
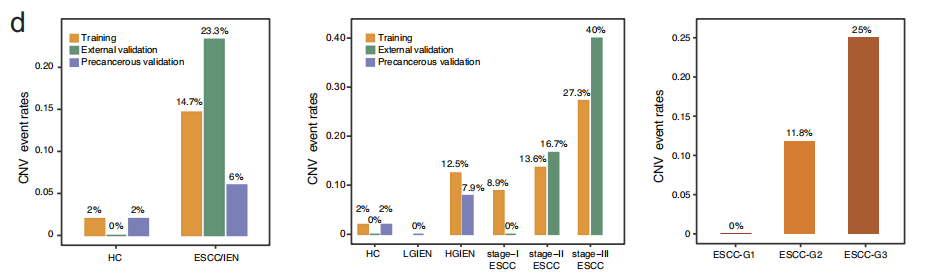
用于ESCC检测的cfDNA CNVs识别。
图片来源:Nature Communications
cfDNA WGBS数据中片段组学
为了进一步揭示cfDNA的多模态特征,作者对发现队列中的cfDNA片段大小进行了全面分析。ESCC患者和HC的cfDNA样本均在166 bp处出现峰值。然而,在ESCC组中观察到更高比例的短片段(90-150 bp)。通过评估cfDNA短片段与人类基因组的比例来计算FSR。ESCC患者和HC在所有bin中的平均FSR没有发现显著差异。然而,ESCC患者中发现了83个FSRs显著升高的bin。在发现队列和外部验证队列中,ESCC患者在这83个选定的bin中的平均FSRs显著更高。但在癌前验证队列的IEN患者中没有观察到这种显著差异(图d)。
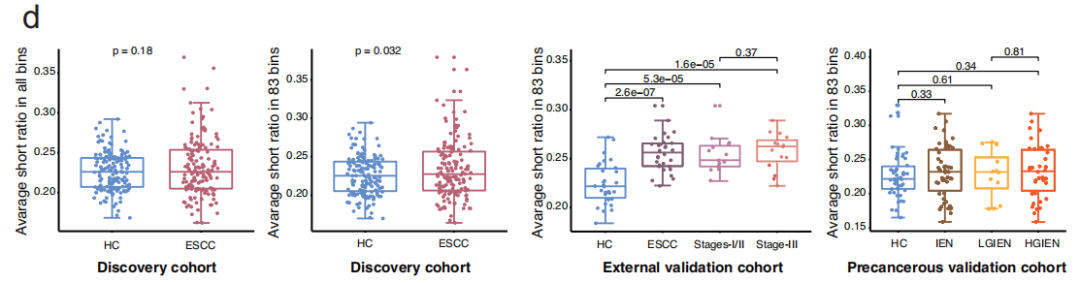
分析WGBS数据中的cfDNA片段的大小分析。
图片来源:Nature Communications
联合EMMA模型对ESCC及癌前病变的准确检测
作者建立了两个联合诊断模型:一个是DMR + CNV模型,另一个是DMR + CNV+FSR模型(EMMA模型)。与ESCC-cfMeth评分相比,DMR + CNV模型与 EMMA模型的表现都显著改善,在外部验证队列(AUC分别为0.94和0.95)和癌前验证队列(AUC均为0.89)中也表现出优异性能(图 c)。当特异性>95%时,EMMA模型在外部验证队列中敏感性为87%,在癌前验证队列中敏感性为62%(图c)。总体而言,EMMA模型将IENs的检出率提高到62%,ESCC一期为78%,二期为83%,III期为93%,同时保持了>95%的特异性。
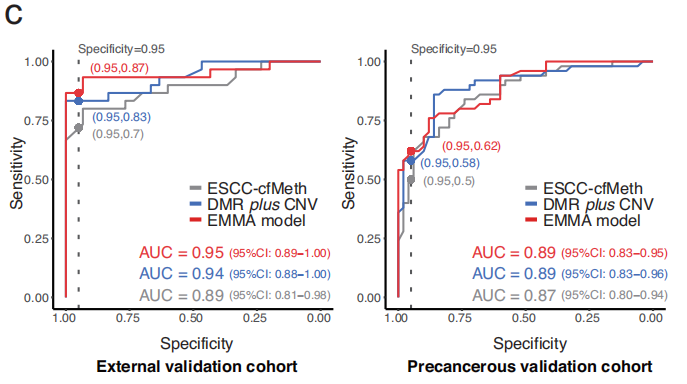
联合EMMA模型对ESCC及癌前病变的准确检测。
图片来源:Nature Communications
总结与讨论
作者对来自非转移性ESCC或癌前病变患者和匹配健康对照的460份cfDNA样本进行了全基因组亚硫酸盐测序(WGBS),并开发了一个扩展的多模态分析(EMMA)框架,以同时识别cfDNA WGBS数据中的cfDNA甲基化、拷贝数变异(CNVs)和片段化特征。cfDNA甲基化标记是最早和最敏感的,在70%的ESCC和50%的癌前病变中可检测到,并且与分子亚型和肿瘤微环境相关。CNVs和片段化特征显示出高特异性,但只与晚期疾病有关。EMMA显著提高了检出率,AUC为0.99,在验证队列中检测到87%的ESCC和62%的癌前病变,特异性>95%。证明了cfDNA甲基组的多模态分析在ESCC早期检测和分子特征监测中的潜力。
但研究也有一些局限性。首先,尽管分析了超过1000个全基因组数据集,但验证队列相对较小。其次,这些多模态标记物的潜在功能仍然未知,特别是在癌前病变中。第三,从WGBS数据中提取CNVs和片段化特征,以节省样本成本。但与cfDNA的WGS数据相比,可能会削弱CNVs的信号和片段特征。第四,突变没有被纳入这个多模态模型。此外,ESCC-cfDNA和EMMA模型的生存益处和预后意义需要在大规模现实世界队列中进一步验证。


